LA SECUENCIACIÓN, UNA HERRAMIENTA CLAVE PARA LA MEDICINA PERSONALIZADA
Hoy en día, gracias al avance de las técnicas de análisis genético podemos seguir de forma sencilla la herencia de variaciones en nuestro genoma. Esto nos ha permitido detectar mutaciones que tienen efectos muy dañinos en el organismo y son la causa de enfermedades o síndromes hereditarios. Pero, estos análisis, además de permitirnos la posibilidad de estudiar y rastrear estas mutaciones de enfermedades hereditarias clásicas, nos permiten generar predicciones de riesgo de enfermedades adquiridas. Conocer los riesgos de padecer una enfermedad cambia el contexto de los términos salud y enfermedad y nos acerca a la medicina personalizada.
Y todo esto lo hemos conseguido gracias a la secuenciación, la técnica que nos permite conocer el orden o secuencia en el que están dispuestas las 4 bases que conforman nuestro ADN (adenina, timina, guanina y citosina) en nuestro genoma o en un fragmento de este. La secuenciación también nos permite conocer la secuencia de nuestro ARN, o del ADN fragmentado o modificado, lo que nos permitirá no solo conocer nuestro genoma, sino también su expresión y su regulación.
¿Pero cómo hemos llegado hasta aquí?, la secuenciación Sanger
En primer lugar, antes de querer asociar cambios en el ADN a enfermedades, surgió el interés de conocer que dice nuestro ADN. Desde que 1869 Friedrich Mietscher aisló el ADN de un organismo, hubo un gran interés en conocer cuál era la secuencia de las bases que formaban esta macromolécula. Durante los años 70 diferentes métodos se pusieron a punto para conocer la secuencia de ARN de trasferencia y Walter Fiers en 1972 sería el primero en secuenciar el ADN de un gen completo. Pero sería Fredrick Sanger el que en 1977 desarrollaría el primer método de secuenciación de ADN valido hasta nuestros días, conocido como secuenciación Sanger. Este método se basa en la replicación del ADN. Pero incluyendo en la reacción dideoxinucleótidos, nucleótidos que carecen del grupo hidroxilo en el carbono 3′, de manera que cuando la polimerasa intenta incorporar un nuevo nucleótido a la hebra de ADN que se está elongando no puede y se detiene. Al detenerse la reacción se formarán cadenas de ADN de diferente longitud, siempre terminando en un dideoxinucleótido, que, si marcamos, podremos conocer si es una adenina, una timina, una guanina o una citosina. Así una vez ordenados los fragmentos por tamaño, podremos ordenar las bases marcadas y ordenarlas a su vez conociendo la secuencia del ADN.
Este método fue mejorado paso a paso a lo largo de los años, obteniendo una mejor detección, eliminado el uso de sustancias radiactivas para su marcaje y posteriormente se consiguió que la fluorescencia emitida por los dideoxinucleótidos fuese adquirida y analizada por computadoras. Todo esto ha hecho que durante 45 años este sea el método más usado para secuenciar el ADN. Ya que es un método que ha permitido leer fragmentos de ADN de hasta 1000pb con un porcentaje de error muy bajo (menor del 0.001% por base), a excepción de zonas complejas con largos homopolímeros.
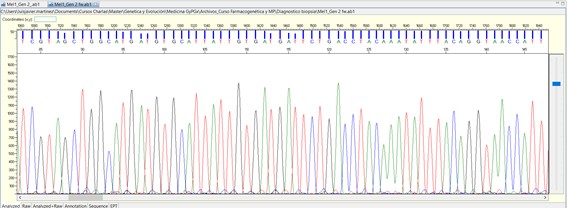
Electrofluorograma resultado de la secuenciación de un fragmento de ADN, donde se pueden ver en cada color cada uno de las bases (A, C, G y T) que lo conforma y su orden.
El tiempo en el que realizaba el análisis de una muestra era aproximadamente 30 minutos, tiempo en el que el ADN recorría un microcapilar tratando de llegar al polo positivo, donde una cámara recogía la fluorescencia del fluorocromo. Algunos equipos leían de forma simultánea 4, 8, 16, 24 o 48 muestras, al tener ese número de capilares, teniendo un rendimiento bastante alto.
Pero esta técnica, en la clínica y sobre todo en la investigación en salud, se ha reemplazado por otro tipo de secuenciación de nueva o segunda generación, NGS por sus siglas en ingles. Técnica que ha disminuido el coste por base y ha permitido estudiar mayor cantidad de genes de una forma mucho más rápida.
Esta técnica, aunque hace una quincena de años, era impensable que reemplazará a la secuenciación Sanger, poco a poco la ha desplazado, para solo usarse en casos concretos.
Secuenciar más y más, la aparición de la secuenciación de nueva generación
La pirosecuenciación fue el germen con el que empezó la secuenciación de nueva generación. Mostafa Ronaghi, Mathias Uhlen y Pȧl Nyŕen en 1996 desarrollaron una nueva técnica basada en realizar la medida de la fluorescencia durante la síntesis de pirofosfato. Gracias a la adición de una enzima degradante de nucleótidos mejoraron la secuenciación por síntesis y la detección de la luminiscencia generada como resultado de la síntesis de pirofosfato. Esto, además lo hicieron de forma totalmente automatizada, por lo que ya se podría definir como una técnica de lectura del ADN de alto rendimiento.
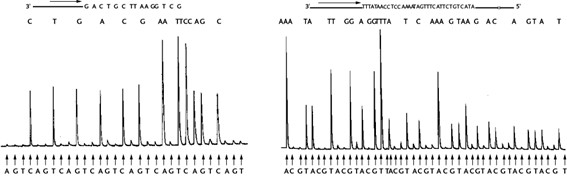
Imagen del resultado de la pirosecuenciación mostrada en el artículo de Mostafa Ronaghi, Mathias Uhlén y Pål Nyrénp publicado en Science en 1998.
Pero no sería hasta 2005 cuando apareciera el primer equipo automatizado de pirosecuenciación. El equipo de Jonathan Rothberg lanzó al mercado la primera plataforma de secuenciación de nueva generación, que implementaba en un equipo compacto la tecnología de pirosecuenciación, el sistema 454.
En paralelo en 1998, David Klenerman y Shankar Balasubramanian desarrollarían un nuevo método de secuenciación por síntesis que utiliza colorantes fluorescentes. Una química de secuenciación que sin saberlo se extendería y se convertiría en la base de la química mas usada para la re-secuenciación en nuestros días. Este método, es la base de la empresa Solexa que fundaron Klenerman y Balasubramanian y que en 2005 obtendría un equipo que generaría más de 3 millones de bases de una sola carrera. La característica clave de su equipo de NGS es la secuenciación paralela masiva de lecturas cortas en una matriz sólida mediante terminadores reversibles. Esta secuenciación masiva en una superficie solida la consiguieron gracias a la miniaturización de las reacciones y la automatización de la carga y lectura.
Dos años después Illumina adquirió la empresa Solexa. Convirtiéndose en poco tiempo en la tecnología NGS más utilizada en el mundo y, hasta hoy, son el líder del mercado de plataformas NGS.
Video 1: Secuenciación por síntesis, química que comenzó a usar el equipo solexa y que actualmente es la marca de identidad de los secuenciadores Illumina.

Imagen del equipo de secuenciación de nueva generación mediante ligasas 5500xl de Life Technologies con su predecesor, el SOLiD 4 a la izquierda de la imagen.
Applied Biosystems optó por otra tecnología, la «secuenciación por ligación», o “secuenciación mediante ligases” del sistema SOLiD en 2007. En esta plataforma se utilizaba la secuenciación por ligación que detectaba fragmentos de PCR generados en una emulsión. Compartían con el equipo 454 la PCR en emulsión y con el solexa la lectura en una fase sólida, pero en este caso, en vez de generarse los fragmentos de ADN en la fase sólida, se adherían a ella los amplicones, previamente unidos a una microesfera. Una vez que todas las esferas estaban en un cristal, se iban añadiendo las diferentes sondas, nucleótidos, dinucleótidos marcados con fluorescencia y soluciones que detectaría la secuencia de cada amplicón. En 2011, Life Technologies (que había adquirido Applied Biosystems), tras su reciente actualización del SOLiD al equipo 5500xl, saco al mercado un equipo de NGS llamado Ion Torrent. Este equipo conservaría la PCR en emulsión pero cambiaría el tipo de detección de los fragmentos amplificados. Los Ion Torrent, al igual que los actuales Proton, aprovecharían el hecho de que la adición de un dNTP a un polímero de ADN libera un ion de hidrógeno para poder detectar este cambio. Usando semiconductores estos equipos son capaces de medir simultáneamente millones de cambios de pH, que generan los iones de hidrógeno liberados. Determinando la secuencia de cada fragmento dependiendo de los cambios en el pH en una micromatriz. Los cambios de pH solo se producirán cuando se una la base complementaria a la interrogada, no con el resto. Esta ha sido la técnica conservada por la casa comercial Life Technologies, actualmente Thermo Fisher Scientific.
En los últimos años ha irrumpido en el mercado una nueva serie de equipos con alta capacidad, que alcanza los 5 mil millones de secuencias por carrera. Puede que finalmente se generalicen en el mercado por la disminución del precio de sus equipos y sus reactivos, lo que se traduce al final en la reducción del coste de la secuenciación. MGI Tech es una compañía que basa sus secuenciadores en una tecnología de secuenciación mediante la introducción de los fragmentos de ADN en esferas, mediante su circularización. Estas esferas de ADN o “nanoball” son las que luego se introducen en sus secuenciadores, la lectura mediante fluorocromos produce lecturas de 150 a 200pb, en lecturas pareadas, leyendo los fragmentos de ADN en ambos sentidos.
Secuenciar fragmentos largos, plataformas especializadas en lecturas largas
En muchas ocasiones, para conocer una secuencia desconocida, una secuencia con muchas repeticiones, discernir entre especies con secuencias similares o ARNs mensajeros que generan isoformas muy similares son necesarias secuencias de gran tamaño. Recordemos que hasta el momento hemos hablado de equipo que secuenciaban hasta 400pb o 600pb si leían una librería por ambos extremos. Debido a esto, se hace patente la necesidad de tecnologías que puedan obtener secuencias que superen los 1000pb.
Han sido fundamentalmente dos empresas las que han ofrecido equipos que su objetivo era este tipo de secuencias Oxford Nanopore Technologies (ONT) y Pacific Biosciences (PacBio). Las plataformas de secuenciación de ONT: MinION, GridION y PromethION y Sequel, Sequel II y Sequel IIe de PacBio generan lecturas con una longitud media de 8 –10 kb y hasta 100 kb. Y si bien en el pasado las tasas de error eran mayores que las de los equipos de Illumina o Thermo Fisher Sc. en los últimos años han ido disminuyendo y mejorando su calidad y rendimiento. Pero el precio de este tipo de secuencias es el repulsivo para su uso generalizado.
Por lo general, cada vez vemos más artículos e investigadores que usan y recomiendan el ensamblaje híbrido, utilizando conjuntos de datos combinados, usando dato de secuenciación de lectura corta y larga. Usando estos las lecturas largas nos dan datos de la estructura del genoma y las cortas facilitan el ensamblaje detallado a escala local, y corrigen los errores en lecturas largas.
Secuenciadores a gusto de todos

Algunos de los equipos disponibles de la casa comercial Illumina, donde se puede ver el número de gigabases y secuencias máximas. Así como la longitud máxima de las secuencias que producen.
Gracias a todos estos avances en la tecnología de secuenciación disponemos en el mercado de muchos y diferentes secuenciadores con diferentes características. Que producen mayor o menor cantidad de secuencias, y estas con diferentes tamaños. Secuencias generadas a precios muy distintos. Equipos que se adaptarán mejor a un proyecto de investigación o a la tarea encomendada a ese equipo. Por tanto, tendremos que saber bien que queremos secuenciar antes de seleccionar el equipo para secuenciarlo.
Solo la casa comercial Illumina dispone en el mercado de 7 equipos de NGS: iSeq 100, MiniSeq, MiSeq, NextSeq 550, NextSeq 1000, NextSeq 2000 y NovaSeq 6000. Estos equipos tienen características muy distintas, van desde producir 4 millones de secuencias en el iSeq 100, hasta 20 mil millones en el Novaseq. Y la longitud de las secuencias producidas varia de 150 pb por cada uno de los extremos de los fragmentos de ADN, en la mayoría de sus eequipos a las 250pb en Novaseq o 300 en Miseq. Y carreras de 4 horas en el MiniSeq a otras de 48 en el NextSeq 2000. Y todos los equipos tienen su utilidad por lo que es buena esa versatilidad. El precio por base secuenciada varia mucho de los equipos con mas capacidad a los mas pequeños variando hasta mas de 10 veces el precio por base secuenciada entre los cartuchos de los equipos mas pequeños (Miseq, Miniseq…) a los precios de Novaseq, donde la base no alcanza los 5×10-9€.
Por tanto, la selección del equipo es muy importante a la hora de abordar un estudio de secuenciación, lo que hace muy difícil que un único laboratorio posea el secuenciador idóneo para una tarea o proyecto. Por lo que hay que hacer una buena selección, para poder realizar el mayor número de proyectos posibles, y, además, cada vez es mejor el trabajo en red entre diferentes centros de secuenciación.
Autor: Dr Luis Javier Martínez González. Responsable de Unidad de Genómica en Genyo (Granada)
Referencias:
2.- A Sequencing Method Based on Real-Time Pyrophosphate. Ronaghi M, Uhlén M, Nyrén P. Science.
3.- Historia de la secuenciación de nueva generación en Illumina.


